Plant Journal |我室胡骏教授课题组揭示了植物细胞器转录组中RNA编辑新位点
来源:admin
浏览量:0
发布时间:2024/01/01
RNA编辑是植物细胞器RNA代谢中广泛存在的重要的转录后修饰过程,其通过C-to-U的碱基修饰保证RNA序列和所翻译蛋白质在进化上的保守性及功能的完整性,对于植物正常的生长发育至关重要。但是,经典的RNA编辑检测方法往往工作量大、准确度低,且十分依赖于特异性引物的设计,这限制了对于植物RNA编辑现象的研究。因此,探索一种快速、准确且适用于多种植物和组织的RNA编辑高通量检测方法对于加速植物RNA编辑的研究是非常重要的。

近日,我室胡骏教授团队在The Plant Journal发表了题为“Profiling of RNA editing events in plant organellar transcriptomes with high-throughput sequencing”的研究论文。该研究分析比较了DSN-seq和Ribo-off-seq两种基于rRNA去除原则的高通量测序方法检测水稻、拟南芥中RNA编辑的效果,并在水稻中成功鉴定到多个新的RNA编辑位点,研究结果表明DSN-seq可作为一种具有重要应用价值的RNA编辑检测方法。
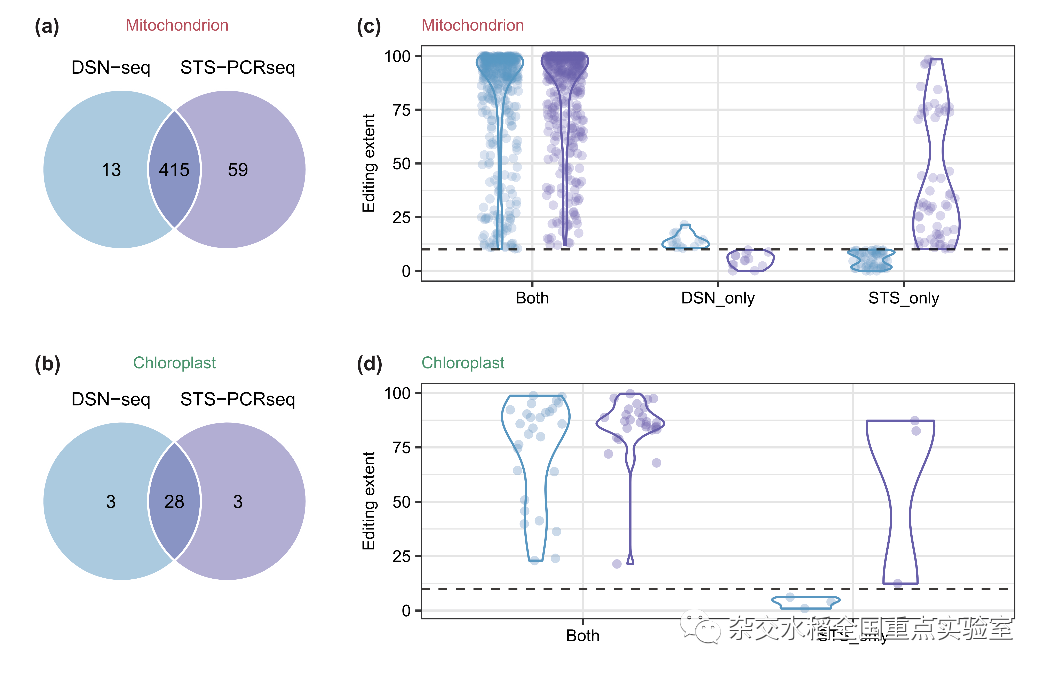
研究结果显示,DSN-seq和Ribo-off-seq都能够有效富集水稻细胞器的转录组信息,而mRNA-seq并不能很好地检测到细胞器转录组的序列。为了对水稻RNA编辑位点进行全面的检测,作者首先结合人工查验和生信分析对现有的水稻细胞器参考基因组信息进行了整合,构建了可靠的参考序列。结果显示,DSN-seq和Ribo-off-seq两种测序方法可以很好地检测已知的RNA编辑位点及其编辑效率。此外,该研究成功在水稻中鉴定到22个新的编辑位点,纠正了4个错误的经典编辑位点,并均利用一代测序进行逐一验证。为后续水稻中RNA编辑的研究提供了可靠的参考信息。
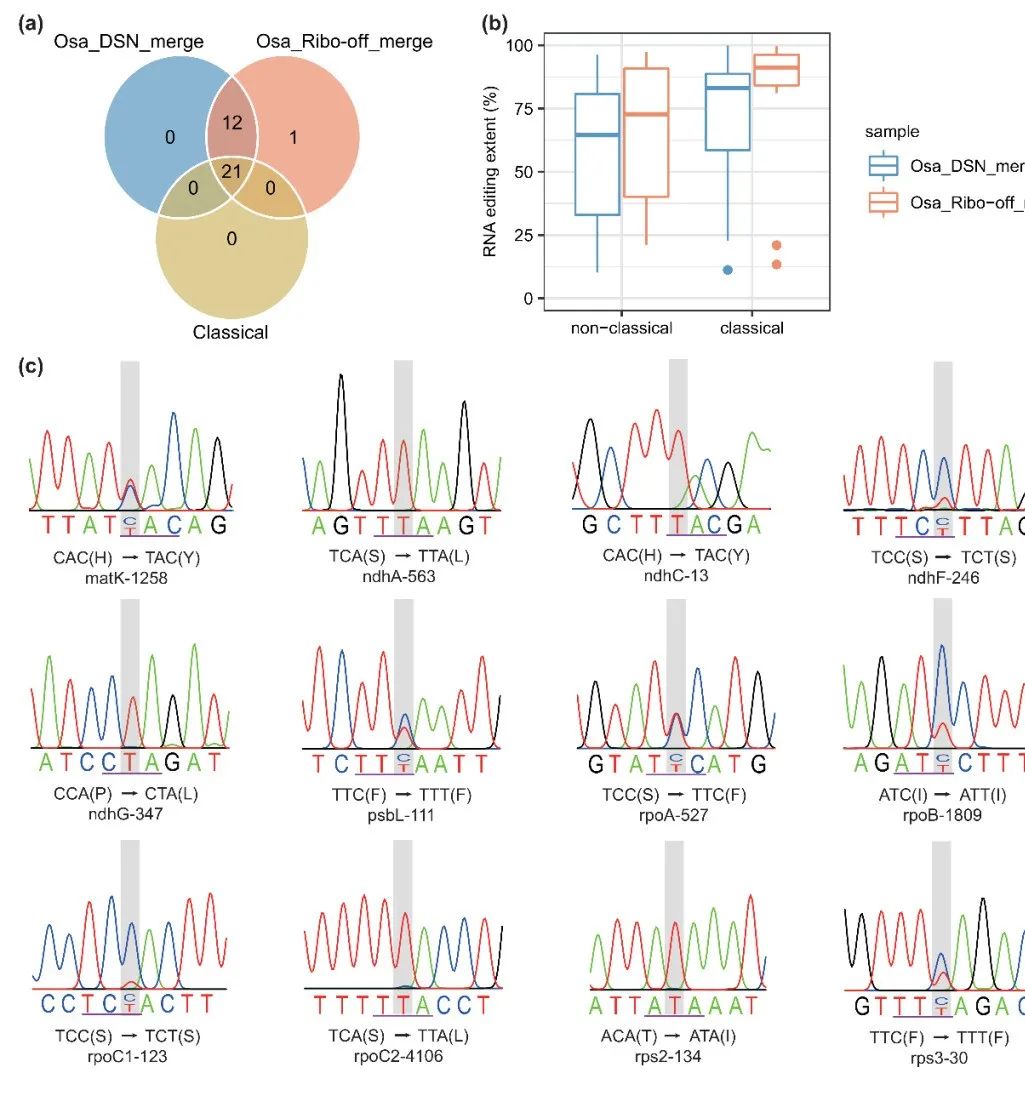
DSN-seq 与 Ribo-off-seq 都展现出了较强的RNA编辑位点检测能力,且DSN-seq 表现出更强的核转录组信息检测能力,对于核质互作研究有重要价值。同时,DSN-seq 在拟南芥中也表现出较好的RNA编辑检测效果,说明DSN-seq在单双子叶植物中都具有良好的应用价值。
综上所述,该研究系统分析了DSN-seq和Ribo-off-seq两种高通量测序方法检测植物RNA编辑位点的效果,获得了更为完整、可靠的水稻RNA编辑图谱。然而,Ribo-off-seq等基于商业化rRNA去除试剂盒的测序方法受到对应试剂盒适用范围的限制,DSN-seq则基于rRNA在总RNA中的超高丰度对其进行去除,并没有序列特异性的限制,这也暗示了DSN-seq这一方法在植物RNA编辑研究中的泛用性价值,为之后基于高通量测序方法的植物RNA编辑研究提供了一定的指导。
杂交水稻全国重点实验室在读博士生刘柯嘉为论文的第一作者,在读博士生谢斌、彭蕾蕾参与材料准备和实验分析,实验室胡骏教授为该论文的通讯作者。武汉康测科技有限公司吴启家为该研究提供技术指导。
该研究得到了国家自然科学基金、湖北省创新群体、杂交水稻全国重点实验室培育项目等资助。
原文链接:https://doi.org/10.1111/tpj.16607
