我室胥国勇教授课题组利用RNA编辑技术鉴定RBP相分离小体中的RNA组分
来源:admin
浏览量:0
发布时间:2021/08/25
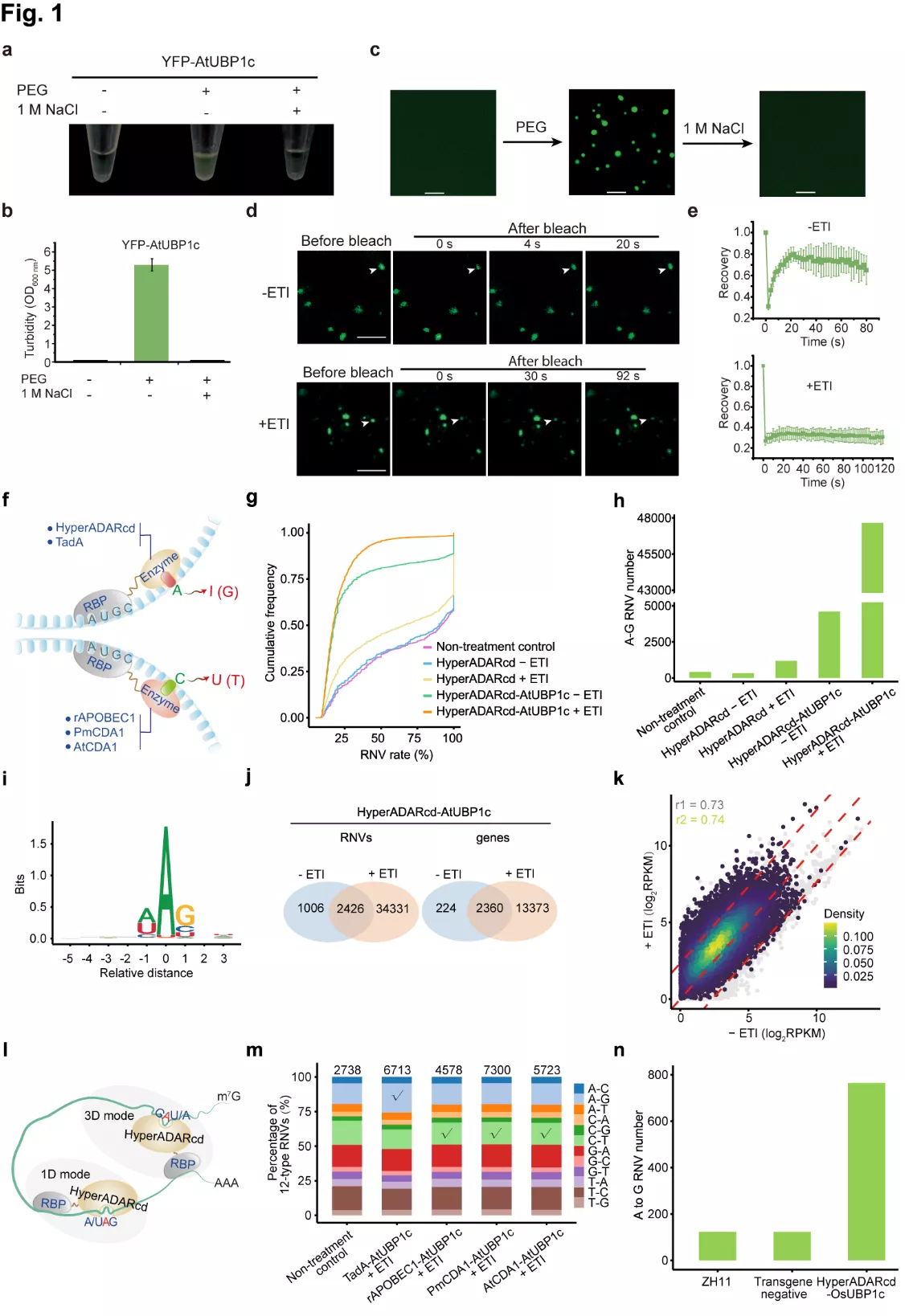
2021年8月24日,我室胥国勇教授课题组在Cell Discovery在线发表了题为Proximity editing to identify RNAs in phase-separated RNA binding protein condensates的研究论文。RNA结合蛋白(RBP)被称作RNA clothes,决定着RNA一生的命运。RBP通常与RNA结合发生相变形成聚集体(phase-separated condensate),从而重编程基因的转录和翻译,但鉴定其中的RNA组分变得十分困难。将改造自线虫的RNA编辑酶HyperADARcd与感兴趣的RBP融合表达,对RBP结合的RNA进行A > G编辑,通过NGS就能实现对RBP相分离小体中RNA的高效鉴定。

RBP和RNA的相互作用确保了遗传信息的正确流动,赋予细胞在时空尺度上发生重编程。拟南芥基因组中存在大量的RBP候选基因(> 6%)【1】,揭示了RBP-RNA相互作用的重要性。然而,许多候选基因被注释为核酸相关功能之外的其他基因(如代谢酶、转运蛋白等),亟需证明这部分基因是真实的RBP,而最直接的方法就是鉴定它们的RNA靶标。尽管传统的紫外交联法结合免疫沉淀(CLIP)是鉴定RNA靶标的标准方法,但目前还没有被广泛应用到植物中。这很可能是因为该方法依赖于高质量的抗体、紫外线交联效率低和操作流程极其复杂【2-4】。这对研究微环境敏感的RBP,以及由于基因突变、发育和环境刺激、体外不相容的实验条件等易于形成聚集体的RBP变得更加困难【5,6】。因此,开发鉴定聚集体中的RNA靶标的方法非常必要。
本研究以拟南芥 AtUBP1c为例,证明了其相分离的细胞行为响应植物免疫信号刺激。通过构建HyperADARcd-AtUBP1c融合蛋白在模式植物烟草和拟南芥中鉴定了免疫信号刺激前后聚集体中的不同RNA组分。研究构建的通用载体和使用的生物信息学pipeline可以分享使用。该系统也成功的应用到了水稻RBP的研究,为其他作物中RBP功能研究提供了可靠的方法借鉴。
我室博士后周贵龙、博士生牛瑞霞和周雨露为本文并列第一作者,胥国勇教授为通讯作者。该研究得到了国家自然科学基金的支持。
参考文献:
1. Köster, T., Marondedze, C., Meyer, K. & Staiger, D. RNA-binding proteins revisited-the emerging Arabidopsis mRNA interactome. Trends Plant Sci. 22,512–526 (2017).
2. Meyer, K. et al. Adaptation of iCLIP to plants determines the binding land-scape of the clock-regulated RNA-binding protein AtGRP7. Genome Biol. 18,204 (2017).
3. Zhang, Y. et al. Integrative genome-wide analysis reveals HLP1, a novel RNA-binding protein, regulates plant flowering by targeting alternative poly-adenylation. Cell Res. 25, 864–876 (2015).
4. Koster, T. & Meyer, K. Plant ribonomics:proteins in search of RNA partners.Trends Plant Sci. 23, 352–365 (2018).
5. Rhine, K., Vidaurre, V. & Myong, S. RNA droplets. Annu. Rev. Biophys. 49,247–265 (2020).
6. Fang, X. et al. Arabidopsis FLL2 promotes liquid–liquid phase separation of polyadenylation complexes. Nature 569, 265–269 (2019).
文章链接: https://doi.org/10.1038/s41421-021-00288-9
https://doi.org/10.1038/s41421-021-00288-9
